Bu değişkenin boş bir array olmasından dolayı hata veriyor. Paylaştığınız örnek veri setinde 4 satır veri olduğu için eğitim için ayıracak pek bir şey kalmıyor ve o da boş oluyor olabilir. İsterseniz örnek veri setindeki veri sayısını artırın (mesela 20-30 gibi).
Öncelikle çok teşekkür ediyorum yardımınız için. Evet dediğiniz gibi verileri arttırmam gerekiyormuş. Aslında aklıma geldi ancak belki başka bir yerde hata var diye düşündüm birden.
Benim bu projedeki amacım elimde Türkiye’de yayınlanan “Ağır Hasta, İyileşen Hasta, Toplam Vaka, Toplam Ölüm Sayısı” gibi verileri alıp işlemek ve tahminde bulunmak. Ben verileri aslında indirdim, hepsini excel’de bir araya getirdim. Bu kısımda benim dikkat etmem gereken konu nedir sizce?
Çalışan projem olunca ikinci aşamada yine LSTM kullanarak diğer ülkelerin verileri ile çalışacağım ama o kısımda hepsini bir arada göstermeye çalışacağım. Ancak bu adımda sadece Türkiye verileri ile çalışmaya çalışıyorum.
Hepsini ayrı ayrı mı tahmin etmeye çalışıyorsunuz yoksa, mesela, Toplam Vaka sayısını diğer 3’ünü de kullanarak mı tahmin etmeye çalışıyorsunuz?
Ağır hasta sayısı verisini kullanarak ölüm oranını tahmin etmek gibi proje değil de. Ölüm oranı için ayrı “Gerçek - Tahmini” grafik ve sonuçlar, Ağır Hasta için “Gerçek - Tahmini” grafik ve sonuçlar gibi.
Sizin yukarıda yaptığınız çalışma aslında uyuyor diye düşünüyorum ama yanlış mıyım bilmiyorum.
Ben her bir veriyi (Ağır Hasta Sayısı, İyileşen Hasta Sayısı, Toplam Vaka Sayısı vs.) ayrı excel çalışması haline getirip csv uzantısı ile kayıt ettim. Daha sonra sizin yukarıda paylaştığınız çalışmayı ayrı ayrı csv uzantılı dosyalar için çalıştıracağım.
Bu şekilde ilerlemem doğru olmuş olur mu sizce?
Evet; ama o kodda validasyon yok tekrar belirteyim, dolayısıyla hiperparametreleri (mesela epoch sayısı, learning rate, LSTM’deki hidden neuron sayısı gibi) seçme kısmını ekleyip kendinize göre düzenlerseniz, bahsettiğiniz amaca hizmet eden bir kod ortaya çıkar diye düşünüyorum.
Ben dün tüm gece bunun ile uğraştım. Biraz okuduğum kitaptan biraz internette yaptığım çalışmalardan bir çalışma tamamladım. Sonuçları elde ettim. Sizinle paylaşmak isterim eğer izniniz olursa. Bu yaptığım çalışma acaba istediğim şeyi yapıyor mu sizce yani bir hatam var mı acaba kod satırlarında. Size danışmak isterim.
Estağfurullah, paylaşabilirsiniz tabii.
Çok teşekkürler paylaşıyorum çalışmamı. Burada hatalı olan yerler acaba nereler sizce?
import math
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from sklearn.preprocessing import MinMaxScaler
from sklearn.metrics import mean_squared_error
from keras.models import Sequential
from keras.layers import Dense, LSTM, Dropout
RANDOM_SEED = 42
TRAIN_CASE = 'agir_hasta'
TRAIN_LOCATION = 'Turkiye'
DATATRAIN_SOURCE_FILE = 'http://facadium.com.tr/lstm_dataset/ornek2/covid19_turkiye_verileri.csv'
TEST_SIZE = 0.3
LOOK_BACK = 1
BATCH_SIZE = 1
EPOCHS = 2000
DAYS_TO_PREDICT = 30
np.random.seed(RANDOM_SEED)
df = pd.read_csv(DATATRAIN_SOURCE_FILE)
if TRAIN_LOCATION:
df = df.loc[df['bolge']==TRAIN_LOCATION]
df.drop(['bolge'], axis=1, inplace=True)
df.set_index('tarih', inplace=True)
df.index = pd.to_datetime(df.index)
df.head()
cases = df.filter([TRAIN_CASE])
cases = cases[(cases.T != 0).any()]
cases.head()
plt.suptitle('Actual Cases Based on \'' + TRAIN_CASE + '\'')
plt.title('bolge = ' + TRAIN_LOCATION, fontsize='medium')
plt.plot(cases, label=TRAIN_CASE)
plt.xlabel('tarih')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
plt.show()
cases.shape
def data_split(data, look_back=1):
x, y = [], []
for i in range(len(data) - look_back - 1):
a = data[i:(i + look_back), 0]
x.append(a)
y.append(data[i + look_back, 0])
return np.array(x), np.array(y)
test_size = TEST_SIZE
test_size = int(cases.shape[0] * test_size)
train_cases = cases[:-test_size]
test_cases = cases[-test_size:]
train_cases.shape
test_cases.shape
scaler = MinMaxScaler(feature_range=(-1, 1))
scaler = scaler.fit(cases)
all_cases = scaler.transform(cases)
train_cases = scaler.transform(train_cases)
test_cases = scaler.transform(test_cases)
all_cases.shape
train_cases.shape
test_cases.shape
look_back = LOOK_BACK
X_all, Y_all = data_split(all_cases, look_back=look_back)
X_train, Y_train = data_split(train_cases, look_back=look_back)
X_test, Y_test = data_split(test_cases, look_back=look_back)
X_all.shape
X_train.shape
X_test.shape
X_all = np.array(X_all).reshape(X_all.shape[0], 1, 1)
Y_all = np.array(Y_all).reshape(Y_all.shape[0], 1)
X_train = np.array(X_train).reshape(X_train.shape[0], 1, 1)
Y_train = np.array(Y_train).reshape(Y_train.shape[0], 1)
X_test = np.array(X_test).reshape(X_test.shape[0], 1, 1)
Y_test = np.array(Y_test).reshape(Y_test.shape[0], 1)
X_all.shape
Y_all.shape
X_train.shape
Y_train.shape
X_test.shape
Y_test.shape
batch_size = BATCH_SIZE
model = Sequential()
model.add(LSTM(4, return_sequences=True,
batch_input_shape=(batch_size, X_train.shape[1], X_train.shape[2]),
stateful=True))
model.add(LSTM(1, stateful=True))
model.add(Dense(Y_train.shape[1]))
model.compile(loss='mean_squared_error', optimizer='adam')
print(model.summary())
epoch = EPOCHS
loss = []
for i in range(epoch):
print('Iteration ' + str(i + 1) + '/' + str(epoch))
model.fit(X_train, Y_train, batch_size=batch_size,
epochs=1, verbose=1, shuffle=False)
h = model.history
loss.append(h.history['loss'][0])
model.reset_states()
plt.plot(loss, label='loss')
plt.title('Model Loss History')
plt.xlabel('epoch')
plt.ylabel('loss')
plt.legend()
plt.show()
train_predict = model.predict(X_train, batch_size=batch_size)
days_to_predict = X_test.shape[0]
test_predict = []
pred_samples = train_predict[-1:]
pred_samples = np.array([pred_samples])
for i in range(days_to_predict):
pred = model.predict(X_test[i:(i+1)], batch_size=batch_size)
pred = np.array(pred).flatten()
test_predict.append(pred)
test_predict = np.array(test_predict).reshape(1, len(test_predict), 1)
model.reset_states()
X_train_flatten = np.array(scaler.inverse_transform(
np.array(X_train).reshape(X_train.shape[0], 1)
)).flatten().astype('int')
Y_train_flatten = np.array(scaler.inverse_transform(
np.array(Y_train).reshape(Y_train.shape[0], 1)
)).flatten().astype('int')
train_predict_flatten = np.array(scaler.inverse_transform(
np.array(train_predict).reshape(train_predict.shape[0], 1)
)).flatten().astype('int')
X_test_flatten = np.array(scaler.inverse_transform(
np.array(X_test).reshape(X_test.shape[0], 1)
)).flatten().astype('int')
Y_test_flatten = np.array(scaler.inverse_transform(
np.array(Y_test).reshape(Y_test.shape[0], 1)
)).flatten().astype('int')
test_predict_flatten = np.array(scaler.inverse_transform(
np.array(test_predict).reshape(test_predict.shape[1], 1)
)).flatten().astype('int')
train_predict_score = math.sqrt(
mean_squared_error(
Y_train_flatten,
train_predict_flatten
)
)
test_predict_score = math.sqrt(
mean_squared_error(
Y_test_flatten,
test_predict_flatten
)
)
'Train Score: %.2f RMSE' % (train_predict_score)
'Test Score: %.2f RMSE' % (test_predict_score)
plt.plot(
cases.index[:len(X_train_flatten)],
X_train_flatten,
label='train (true)'
)
plt.plot(
cases.index[:len(train_predict_flatten)],
train_predict_flatten,
label='train (predict)'
)
plt.plot(
cases.index[len(X_train_flatten):len(X_train_flatten) + len(X_test_flatten)],
X_test_flatten,
label='test (true)'
)
plt.plot(
cases.index[len(X_train_flatten):len(X_train_flatten) + len(test_predict_flatten)],
test_predict_flatten,
label='test (predict)'
)
plt.suptitle('Historical Training Test Based on \'' + TRAIN_CASE + '\'')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
model.reset_states()
epoch = EPOCHS
loss = []
for i in range(epoch):
print('Iteration ' + str(i + 1) + '/' + str(epoch))
model.fit(X_all, Y_all, batch_size=batch_size,
epochs=1, verbose=1, shuffle=False)
h = model.history
loss.append(h.history['loss'][0])
model.reset_states()
plt.plot(loss, label='loss')
plt.title('Model Loss History')
plt.xlabel('epoch')
plt.ylabel('loss')
plt.legend()
plt.show()
all_predict = model.predict(X_all, batch_size=batch_size)
days_to_predict = DAYS_TO_PREDICT
future_predict = []
pred_samples = all_predict[-1:]
pred_samples = np.array([pred_samples])
for i in range(days_to_predict):
pred = model.predict(pred_samples, batch_size=batch_size)
pred = np.array(pred).flatten()
future_predict.append(pred)
new_samples = np.array(pred_samples).flatten()
new_samples = np.append(new_samples, [pred])
new_samples = new_samples[1:]
pred_samples = np.array(new_samples).reshape(1, 1, 1)
future_predict = np.array(future_predict).reshape(len(future_predict), 1, 1)
model.reset_states()
f_future_predict = model.predict(future_predict, batch_size=batch_size)
model.reset_states()
X_all_flatten = np.array(scaler.inverse_transform(
np.array(X_all).reshape(X_all.shape[0], 1)
)).flatten().astype('int')
X_all_flatten = np.absolute(X_all_flatten)
Y_all_flatten = np.array(scaler.inverse_transform(
np.array(Y_all).reshape(Y_all.shape[0], 1)
)).flatten().astype('int')
Y_all_flatten = np.absolute(Y_all_flatten)
all_predict_flatten = np.array(scaler.inverse_transform(
np.array(all_predict).reshape(all_predict.shape[0], 1)
)).flatten().astype('int')
all_predict_flatten = np.absolute(all_predict_flatten)
future_predict_flatten = np.array(scaler.inverse_transform(
np.array(future_predict).reshape(future_predict.shape[0], 1)
)).flatten().astype('int')
future_predict_flatten = np.absolute(future_predict_flatten)
f_future_predict_flatten = np.array(scaler.inverse_transform(
np.array(f_future_predict).reshape(f_future_predict.shape[0], 1)
)).flatten().astype('int')
f_future_predict_flatten = np.absolute(f_future_predict_flatten)
all_predict_score = math.sqrt(
mean_squared_error(
Y_all_flatten,
all_predict_flatten
)
)
'All Score: %.2f RMSE' % (all_predict_score)
future_index = pd.date_range(start=cases.index[-1], periods=days_to_predict + 1, closed='right')
plt.plot(
future_index,
future_predict_flatten,
label='prediction cases'
)
plt.suptitle('Future Prediction Based on \'' + TRAIN_CASE + '\'')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
plt.plot(
future_index,
f_future_predict_flatten,
label='f_prediction cases'
)
plt.suptitle('F_Future Prediction Based on Previous Future Prediction')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
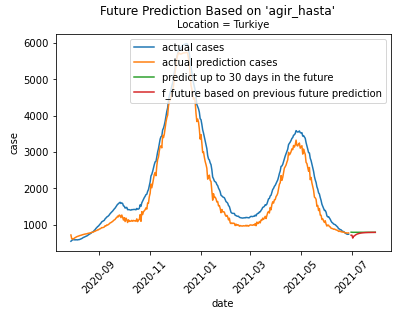
plt.plot(
cases.index[:len(X_all_flatten)],
X_all_flatten,
label='actual cases'
)
plt.plot(
cases.index[:len(X_all_flatten)],
all_predict_flatten,
label='actual prediction cases'
)
plt.plot(
future_index,
future_predict_flatten,
label='predict up to ' + str(days_to_predict) + ' days in the future'
)
plt.plot(
future_index,
f_future_predict_flatten,
label='f_future based on previous future prediction'
)
plt.suptitle('Future Prediction Based on \'' + TRAIN_CASE + '\'')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
Elinize sağlık. Şurada bir problem var:
Test verisi scale edilirken test verisinin istatistiği de kullanılıyor. Yani scaler.fit(cases) dediğinizde, cases test verilerini de içerdiğinden, daha önceden hiç görmediğimizi varsaymamız gereken, modelin genelleyebilirliğini ölçmeye yarayan test setinden istatistik sızmış oluyor. Bu da modelin test verisinde olağandan daha iyi bir sonuç vermesini sağlayabilir.
Bunun önüne geçmek adına scaler yalnız training verisine fit edilmelidir, sonrasında hem training hem de test verisini transform etmelidir. Böylelikle sadece eğitim verisinin istatistiklerini (mesela burada min/max) kullanmış oluruz, sızıntı olmaz, modelin güvenilirliği artar.
Dikkat ederseniz yukarıdaki kodda bu şekilde yapıyoruz:
fit_transform metodu ayrı ayrı fit ve transform yazmaya eşdeğerdir (ama daha hızlıdır takdir edersiniz ki). Önemli olan husus, scaler_X’in yalnız train_X verisi üzerinden fit edilmiş olması. Sonrasında hem train_X’i hem de test_X’i transform eder.
Yani sizdeki kodu şöyle değiştirmek icap eder:
scaler = MinMaxScaler(feature_range=(-1, 1))
# scaler = scaler.fit(cases) # problemli satır
# all_cases = scaler.transform(cases) # burayı tam anlayamadım, aşağıda soruyorum.
# 1. Seçenek: `fit` ve `transform` ayrı
scaler.fit(train_cases)
train_cases = scaler.transform(train_cases)
test_cases = scaler.transform(test_cases)
# 2. Seçenek: `fit_transform` ile 1 satırdan tasarruf
# (operasyonel olarak 1. Seçenekten farkı yok; size kalmış)
train_cases = scaler.fit_transform(train_cases)
test_cases = scaler.transform(test_cases)
Bunun haricinde all_cases’ın olayını tam anlayamadım. Kodda da anladığım kadarıyla 2 ayrı training loop’unuz var:
1.si burası:
2.si ise şurası:
Birinde X_train kullanıyorsunuz diğerinde X_all. Benim anlamadığım bu ikinci kısım; neden training/test ayrımının yanı sıra tüm veriye de fit ediyorsunuz?
Son olarak da yukarıda da bahsedildiği üzere bu akışa bir de validasyonun eklenmesi gerekir. Hiperparametre dediğimiz, makine tarafından öğrenilmeyen ama kullanıcı tarafından elle belirlenmesi gereken parametrelerin seçilme mevzusu. Bunlara örnek olarak kaç gün geriye bakılacağı, optimizer’ın learning rate’i (hatta optimizer’ın kendisi…), LSTM’in kaç layer’dan oluşacağı, LSTM’in layer(lar)ında kaç tane hidden neuron bulunacağı, epoch sayısı, batch size… Bunları yine veriyi kullanarak seçmek gerekir. Burada en az 2 yol akla geliyor: training verisini training + validation diye ayırmak, veya cross-validation yapmak. Bunları da göz önünde bulundurmak isteyebilirsiniz.
En son olarak da math.sqrt(mean_squared_error(...)) yerine mean_squared_error(..., squared=False)'u kullanabilirsiniz.
Hocam merhabalar tekrardan. Ben yazdığınızdan beri inceliyorum ve çalışmalar yapıyorum. Ancak tam çözümünü bulamadım. Acaba bu sorunları nasıl çözebilirim? Bu konuda yardımcı olabilir misiniz acaba?
Merhaba,
1. mevzu: Scaler’ın (StandardScaler) yalnız ve yalnız training verisine fit edilmesi gerekir; sonrasında hem training hem de test verisi transform edilir. Bu çözüm yukarıda paylaşıldığı gibi
# training -> fit & transform
# test -> transform
train_cases = scaler.fit_transform(train_cases)
test_cases = scaler.transform(test_cases)
Bu aslında çok kritik bir durum. Test verisi modelin genelleme yeteneğenin bir ölçüsü olduğundan, yani tabir-i caizse vahşi doğada nasıl davranacağının bir proxy’si olması gerektiğinden, test verisinden hiçbir sızıntı yapılmaması gerekir, bu test verisinin ortalaması/standart sapması olsa dahi. Alttaki 2. mevzu da buraya biraz dokunuyor.
2. mevzu: Model, veriye veri training ve test diye ayrıldıktan sonra, training kısmına fit edilmelidir. Verinin tümüne fit etmek test verisinin amacını baltalar (genellemeyi ölçemeyiz). Bunu all_cases'a fit ederken yapıyorsunuz, çözümü o kısımları kaldırmaktır. Bu kısımları yukarıdaki mesajda belirttim, dikkatinize sunarım.
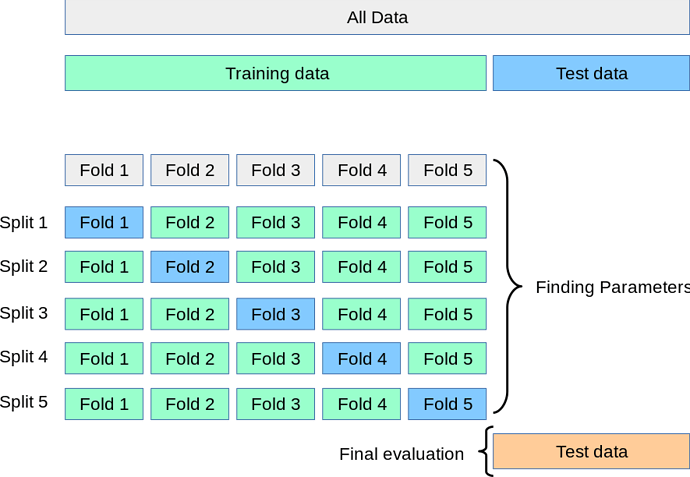
3. mevzu: Hiperparametrelerin ayarlanması. Yukarıdaki hiperparametrelerin neler olduğundan ve öneminden bahsetmeye çalıştım. Bunu yapmanın birkaç yolu da vardır: mesela cross validation, veya 3. bir veri ayrımı olarak validation verisi. Cross validation’dan bahsedelim. Bu da yapılış tarzı bakımından birkaç kısma ayrılır; biz en yaygın olan K-Fold cross validation’dan bahsedelim. Burada aranacak hiperparametrelerin seçilmesi hususunda da birkaç yol vardır: Grid search, Randomized search, Bayesian search gibi. Grid search’ten bahsedelim.
Mevzuyu aktarabilmek adına sizinkinden daha basit bir ağ düşünelim: 1 hidden layer’ı olan bir MLP olsun. Yine regression hedefliyoruz sizdeki gibi.
Diyelim ki 2 hiperparametreyi tune etmek istiyoruz: kaç epoch eğitim yapılacağı ve hidden layer’da kaç adet neuron olacağı. (not: bunlar öğrenilebilir parametreler değildir weight’lerin aksine; bunları kullanıcının elle seçmesi gerekir. Bu amaca binaen cross-validation’a başvuruyoruz.) Bu parametrelerin de denenmesini istediğiniz bazı değerleri vardır, onları kaydediyoruz.
hiperparametre denensin istediğiniz değerler
-------------- -----------------------------
epoch_sayısı 50, 150, 400
hidden_neuron_sayısı 20, 30
Burada epoch için 3, hidden neuron sayısı için 2 değer belirledik (örnek olarak). Grid-search demiştik ya, onun yaptığı tüm kombinasyonlara (grid’e) bakmak oluyor. Burada 2 x 3 = 6 kombinasyon var: [(50, 20), (50, 30), (150, 20), (150, 30), (400, 20), (400, 30)]. Hepsini deneyip “en iyi” olanı seçeceğiz. Neye göre en iyi?
Cross validation söylüyor onu. scikit-learn’den şu görseli aşırdım:
K-fold demiştik ya: burada K = 5. Training verisi 5 parçaya ayrılıyor. Sonra bu ayrım üzerinden K = 5 kere geçiliyor. Her geçişte 4 fold training olarak kullanılıyor, o 4 yerde eğitilen model geride kalan 5. fold’da test ediliyor. Bu “test” fold’u her dönüşte değişiyor (Fold 1, Fold 2, … açık mavi ile, diagonal’da). Sonra bu 5 dönüşteki elde edilen “test” performanslarının ortalaması alınıyor. Bu işlemler "grid"deki her kombinasyon için yapılıyor (bizde 6 tane var). Sonrasında her kombinasyonun bir performansı oluyor o test fold’larının ortalamasından elde edilen. Hangisininki en iyiyse o kombinasyon en iyidir, onu seçeriz. Mesela skorlar sırasıyla [23.4, 61.7, 19.9, 28.8, 44.4, 57.0] çıktı diyelim (artık metrik ne ise [mesela RMSE olabilir]). Bunlar arasında en iyi olan en az RMSE’yi veren, yani 19.9’u veren (150, 20) kombinasyonudur. Sonuca varıyoruz: epoch sayısı 150 olacak, hidden neuron sayısı da 20.
Şimdi ise tüm training verisine (şekilde en üstte “Training data” diye belirtilen, bizim fold’lara böldüğümüz veriye) geri dönüyoruz. epoch = 150, hidden_neuron = 20 diye fiksleyip eğitimi gerçekleştiriyoruz.
En son ise modelin performansını ölçmek adına, şu ana kadar hiç dokunmadığımız test verisine gideriz, orada tahminleri elde edip, RMSE’yi bildiririz; bu modelin performansı budur denilir.
Bu cross-validation mevzusunun kodunu keras’ta scikit learn wrapper’ı ile yazabiliriz. Bir örnek paylaşayım.
import numpy as np
from sklearn.datasets import make_friedman1
from sklearn.metrics import mean_squared_error
from sklearn.model_selection import GridSearchCV, train_test_split
from sklearn.preprocessing import StandardScaler
from tensorflow.keras import Sequential
from tensorflow.keras.layers import Dense
from tensorflow.keras.wrappers.scikit_learn import KerasRegressor
# örnek bir veri
N_SAMPLES = 400
N_FEATURES = 5
X_all, y_all = make_friedman1(n_samples=N_SAMPLES, n_features=N_FEATURES)
# training-test ayrımı
X_train, X_test, y_train, y_test = train_test_split(X_all, y_all,
test_size=0.2)
# scaling: X (train -> fit & transform; test -> transform)
scaler_X = StandardScaler()
X_train = scaler_X.fit_transform(X_train)
X_test = scaler_X.transform(X_test)
# scaling: y (scaler 2D beklediğinden `np.newaxis` ile 2D'ye dönüştürüyoruz;
# geri dönen yine 2D, onu da `ravel` ile 1D'ye tekrar döndürüyoruz...)
scaler_y = StandardScaler()
y_train = scaler_y.fit_transform(y_train[:, np.newaxis]).ravel()
y_test = scaler_y.transform(y_test[:, np.newaxis]).ravel()
# Keras'ı sklearn ile buluşturmak için bu fonksiyon
def make_model(num_hidden_neurons):
# modelimiz
model = Sequential()
# 1 hidden layer var; `num_hidden_neurons`'u tune edeceğiz, hatırlatayım
# Aktivasyon olarak da ReLU tercih edelim (fix o; tune etmiyoruz)
model.add(Dense(num_hidden_neurons,
input_dim=N_FEATURES,
activation="relu"))
# Output layer'ı; 1 tek sayı amaçlıyoruz; activation "linear" varsayılandır
# aslında ama yine de açıklık için yazıyoruz; f(x) = x
model.add(Dense(1,
activation="linear"))
# Derliyoruz
model.compile(optimizer="sgd", loss="mse")
return model
# Yukarıdaki model'i sklearn'e "wrap" ederiz; sebebi sklearn'deki cross-validat
# ion teknolojilerinden yararlanmak :)
model = KerasRegressor(build_fn=make_model)
# "Grid" `"epochs"` Keras tarafından bilinen bir parametre.
# `"num_hidden_neurons"` ise değil; bunu fonksiyonun parametrelerinin isminden
# çıkarıyor (`make_model` fonksiyonunun); o yüzden o konuda tutarlılık gerekir
epochs = [50, 150, 400]
num_hidden_neurons = [20, 30]
param_grid = {"epochs": epochs, "num_hidden_neurons": num_hidden_neurons}
# Grid search ve K-Fold ile Cross validation
K = 5
grid_cv = GridSearchCV(estimator=model, param_grid=param_grid,
cv=K, # K-fold'un K'sı; üstteki şekildeki 5 fold ayrımı
scoring="neg_mean_squared_error") # "en iyi" metriği
grid_cv.fit(X_train, y_train)
# Validasyon tamamlandı. "En iyi" parametreleri elde edelim incelemek adına
best_params_cv = grid_cv.best_params_
print(f"Cross Validation ile bulunan en iyi parametreler: {best_params_cv}")
# Şimdi "en iyi" modelle tüm training verisini hedefleriz
best_model = grid_cv.best_estimator_ # en iyi parametreler bunun içerisinde
best_model.fit(X_train, y_train)
# Test verisinde tahmin yapabiliriz artık
preds_test_raw = best_model.predict(X_test)
# Performansı ölçelim; y'leri başta scale ettiğimiz için geri scale etmek
# gerekir aynı scaler ile (`scaler_y` ile)
preds_test = scaler_y.inverse_transform(preds_test_raw[:, np.newaxis]).ravel()
rmse_model = mean_squared_error(y_test, preds_test, squared=False)
print("Modelin bu veri üzerindeki Test verisi performansı "
f"{rmse_model:.3f} RMSE")
Hocam merhabalar,
Ben dediklerinizi uygulamaya çalışıyorum ancak sürekli bir yerlerde hata alıyorum. En sonunda yaptığımı sandım ancak bu seferde aldığım çıktılar bana hatalı geldi. Acaba bu kodlarda nerede hata yapıyorum? Yardımcı olur musun lütfen?
import math
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from sklearn.preprocessing import MinMaxScaler
from sklearn.metrics import mean_squared_error
from keras.models import Sequential
from keras.layers import Dense, LSTM, Dropout
RANDOM_SEED = 42
TRAIN_CASE = 'agir_hasta'
TRAIN_LOCATION = 'Turkiye'
DATATRAIN_SOURCE_FILE = 'http://facadium.com.tr/lstm_dataset/ornek2/covid19_turkiye_verileri.csv'
TEST_SIZE = 0.3
LOOK_BACK = 1
BATCH_SIZE = 1
EPOCHS = 2000
DAYS_TO_PREDICT = 30
np.random.seed(RANDOM_SEED)
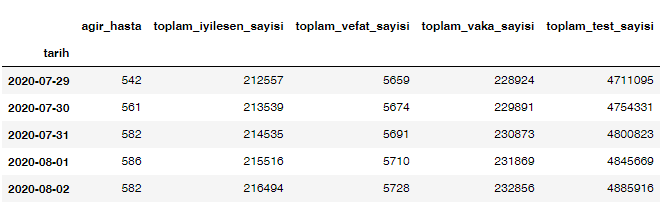
df = pd.read_csv(DATATRAIN_SOURCE_FILE)
if TRAIN_LOCATION:
df = df.loc[df['bolge']==TRAIN_LOCATION]
df.drop(['bolge'], axis=1, inplace=True)
df.set_index('tarih', inplace=True)
df.index = pd.to_datetime(df.index)
df = pd.read_csv(DATATRAIN_SOURCE_FILE)
if TRAIN_LOCATION:
df = df.loc[df['bolge']==TRAIN_LOCATION]
df.drop(['bolge'], axis=1, inplace=True)
df.set_index('tarih', inplace=True)
df.index = pd.to_datetime(df.index)
df.head()
cases = df.filter([TRAIN_CASE])
cases = cases[(cases.T != 0).any()]
cases.head()
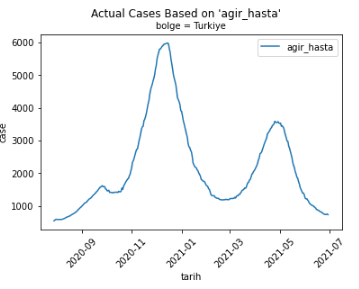
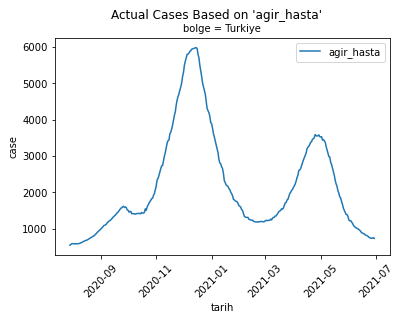
plt.suptitle('Actual Cases Based on \'' + TRAIN_CASE + '\'')
plt.title('bolge = ' + TRAIN_LOCATION, fontsize='medium')
plt.plot(cases, label=TRAIN_CASE)
plt.xlabel('tarih')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
plt.show()
cases.shape
![]()
def data_split(data, look_back=1):
x, y = [], []
for i in range(len(data) - look_back - 1):
a = data[i:(i + look_back), 0]
x.append(a)
y.append(data[i + look_back, 0])
return np.array(x), np.array(y)
test_size = TEST_SIZE
test_size = int(cases.shape[0] * test_size)
train_cases = cases[:-test_size]
test_cases = cases[-test_size:]
train_cases.shape
![]()
test_cases.shape
![]()
scaler = MinMaxScaler(feature_range=(-1, 1))
# scaler = scaler.fit(cases) # problemli satır
# all_cases = scaler.transform(cases) # burayı tam anlayamadım, aşağıda soruyorum.
# 1. Seçenek: `fit` ve `transform` ayrı
scaler.fit(train_cases)
train_cases = scaler.transform(train_cases)
test_cases = scaler.transform(test_cases)
train_cases.shape
![]()
test_cases.shape
![]()
scaler = MinMaxScaler(feature_range=(-1, 1))
scaler = scaler.fit(cases)
all_cases = scaler.transform(cases)
train_cases = scaler.transform(train_cases)
test_cases = scaler.transform(test_cases)
all_cases.shape
![]()
train_cases.shape
![]()
test_cases.shape
![]()
look_back = LOOK_BACK
X_all, Y_all = data_split(all_cases, look_back=look_back)
X_train, Y_train = data_split(train_cases, look_back=look_back)
X_test, Y_test = data_split(test_cases, look_back=look_back)
X_all.shape
![]()
X_train.shape
![]()
X_test.shape
![]()
X_all = np.array(X_all).reshape(X_all.shape[0], 1, 1)
Y_all = np.array(Y_all).reshape(Y_all.shape[0], 1)
X_train = np.array(X_train).reshape(X_train.shape[0], 1, 1)
Y_train = np.array(Y_train).reshape(Y_train.shape[0], 1)
X_test = np.array(X_test).reshape(X_test.shape[0], 1, 1)
Y_test = np.array(Y_test).reshape(Y_test.shape[0], 1)
X_all.shape
![]()
Y_all.shape
![]()
X_train.shape
![]()
Y_train.shape
![]()
X_test.shape
![]()
Y_test.shape
![]()
batch_size = BATCH_SIZE
model = Sequential()
model.add(LSTM(4, return_sequences=True,
batch_input_shape=(batch_size, X_train.shape[1], X_train.shape[2]),
stateful=True))
model.add(LSTM(1, stateful=True))
model.add(Dense(Y_train.shape[1]))
model.compile(loss='mean_squared_error', optimizer='adam')
print(model.summary())

epoch = EPOCHS
loss = []
for i in range(epoch):
print('Iteration ' + str(i + 1) + '/' + str(epoch))
model.fit(X_train, Y_train, batch_size=batch_size,
epochs=1, verbose=1, shuffle=False)
h = model.history
loss.append(h.history['loss'][0])
model.reset_states()

plt.plot(loss, label='loss')
plt.title('Model Loss History')
plt.xlabel('epoch')
plt.ylabel('loss')
plt.legend()
plt.show()
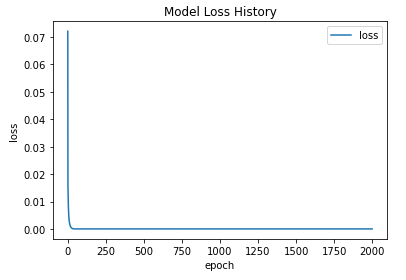
train_predict = model.predict(X_train, batch_size=batch_size)
days_to_predict = X_test.shape[0]
test_predict = []
pred_samples = train_predict[-1:]
pred_samples = np.array([pred_samples])
for i in range(days_to_predict):
pred = model.predict(X_test[i:(i+1)], batch_size=batch_size)
pred = np.array(pred).flatten()
test_predict.append(pred)
test_predict = np.array(test_predict).reshape(1, len(test_predict), 1)
model.reset_states()
X_train_flatten = np.array(scaler.inverse_transform(
np.array(X_train).reshape(X_train.shape[0], 1)
)).flatten().astype('int')
Y_train_flatten = np.array(scaler.inverse_transform(
np.array(Y_train).reshape(Y_train.shape[0], 1)
)).flatten().astype('int')
train_predict_flatten = np.array(scaler.inverse_transform(
np.array(train_predict).reshape(train_predict.shape[0], 1)
)).flatten().astype('int')
X_test_flatten = np.array(scaler.inverse_transform(
np.array(X_test).reshape(X_test.shape[0], 1)
)).flatten().astype('int')
Y_test_flatten = np.array(scaler.inverse_transform(
np.array(Y_test).reshape(Y_test.shape[0], 1)
)).flatten().astype('int')
test_predict_flatten = np.array(scaler.inverse_transform(
np.array(test_predict).reshape(test_predict.shape[1], 1)
)).flatten().astype('int')
train_predict_score = math.sqrt(
mean_squared_error(
Y_train_flatten,
train_predict_flatten
)
)
test_predict_score = math.sqrt(
mean_squared_error(
Y_test_flatten,
test_predict_flatten
)
)
'Train Score: %.2f RMSE' % (train_predict_score)
![]()
'Test Score: %.2f RMSE' % (test_predict_score)
![]()
plt.plot(
cases.index[:len(X_train_flatten)],
X_train_flatten,
label='train (true)'
)
plt.plot(
cases.index[:len(train_predict_flatten)],
train_predict_flatten,
label='train (predict)'
)
plt.plot(
cases.index[len(X_train_flatten):len(X_train_flatten) + len(X_test_flatten)],
X_test_flatten,
label='test (true)'
)
plt.plot(
cases.index[len(X_train_flatten):len(X_train_flatten) + len(test_predict_flatten)],
test_predict_flatten,
label='test (predict)'
)
plt.suptitle('Historical Training Test Based on \'' + TRAIN_CASE + '\'')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
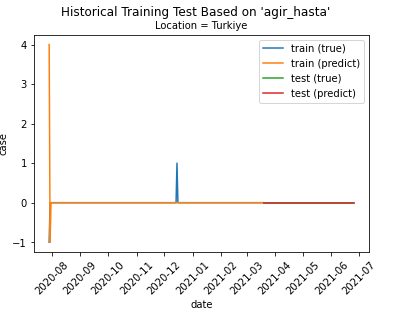
model.reset_states()
epoch = EPOCHS
loss = []
for i in range(epoch):
print('Iteration ' + str(i + 1) + '/' + str(epoch))
model.fit(X_all, Y_all, batch_size=batch_size,
epochs=1, verbose=1, shuffle=False)
h = model.history
loss.append(h.history['loss'][0])
model.reset_states()

plt.plot(loss, label='loss')
plt.title('Model Loss History')
plt.xlabel('epoch')
plt.ylabel('loss')
plt.legend()
plt.show()
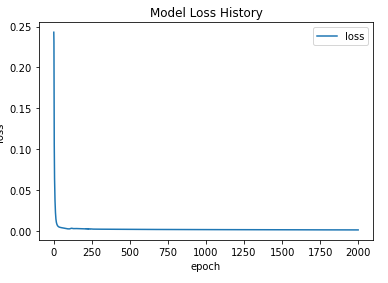
all_predict = model.predict(X_all, batch_size=batch_size)
days_to_predict = DAYS_TO_PREDICT
future_predict = []
pred_samples = all_predict[-1:]
pred_samples = np.array([pred_samples])
for i in range(days_to_predict):
pred = model.predict(pred_samples, batch_size=batch_size)
pred = np.array(pred).flatten()
future_predict.append(pred)
new_samples = np.array(pred_samples).flatten()
new_samples = np.append(new_samples, [pred])
new_samples = new_samples[1:]
pred_samples = np.array(new_samples).reshape(1, 1, 1)
future_predict = np.array(future_predict).reshape(len(future_predict), 1, 1)
model.reset_states()
f_future_predict = model.predict(future_predict, batch_size=batch_size)
model.reset_states()
X_all_flatten = np.array(scaler.inverse_transform(
np.array(X_all).reshape(X_all.shape[0], 1)
)).flatten().astype('int')
X_all_flatten = np.absolute(X_all_flatten)
Y_all_flatten = np.array(scaler.inverse_transform(
np.array(Y_all).reshape(Y_all.shape[0], 1)
)).flatten().astype('int')
Y_all_flatten = np.absolute(Y_all_flatten)
all_predict_flatten = np.array(scaler.inverse_transform(
np.array(all_predict).reshape(all_predict.shape[0], 1)
)).flatten().astype('int')
all_predict_flatten = np.absolute(all_predict_flatten)
future_predict_flatten = np.array(scaler.inverse_transform(
np.array(future_predict).reshape(future_predict.shape[0], 1)
)).flatten().astype('int')
future_predict_flatten = np.absolute(future_predict_flatten)
f_future_predict_flatten = np.array(scaler.inverse_transform(
np.array(f_future_predict).reshape(f_future_predict.shape[0], 1)
)).flatten().astype('int')
f_future_predict_flatten = np.absolute(f_future_predict_flatten)
all_predict_score = math.sqrt(
mean_squared_error(
Y_all_flatten,
all_predict_flatten
)
)
'All Score: %.2f RMSE' % (all_predict_score)
![]()
future_index = pd.date_range(start=cases.index[-1], periods=days_to_predict + 1, closed='right')
plt.plot(
future_index,
future_predict_flatten,
label='prediction cases'
)
plt.suptitle('Future Prediction Based on \'' + TRAIN_CASE + '\'')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
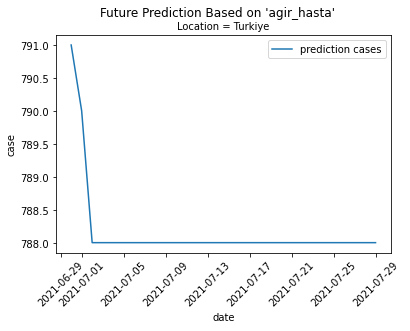
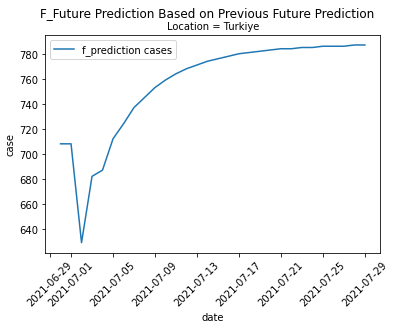
plt.plot(
future_index,
f_future_predict_flatten,
label='f_prediction cases'
)
plt.suptitle('F_Future Prediction Based on Previous Future Prediction')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()
plt.plot(
cases.index[:len(X_all_flatten)],
X_all_flatten,
label='actual cases'
)
plt.plot(
cases.index[:len(X_all_flatten)],
all_predict_flatten,
label='actual prediction cases'
)
plt.plot(
future_index,
future_predict_flatten,
label='predict up to ' + str(days_to_predict) + ' days in the future'
)
plt.plot(
future_index,
f_future_predict_flatten,
label='f_future based on previous future prediction'
)
plt.suptitle('Future Prediction Based on \'' + TRAIN_CASE + '\'')
plt.title('Location = ' + TRAIN_LOCATION, fontsize='medium')
plt.xlabel('date')
plt.ylabel('case')
plt.xticks(rotation=45)
plt.legend()
plt.show()